Nature | 单细胞大模型Geneformer:迁移学习开启基因网络预测新时代
Geneformer是首个基于单细胞转录组的预训练迁移学习模型。
欢迎大家关注 微信公众号|计算生物前沿
发表在Nature上的重大研究提出Geneformer——基于3000万单细胞转录组预训练的深度学习模型,通过迁移学习实现在少量数据下精准预测基因调控网络。该模型能识别剂量敏感致病基因,预测染色质动态,解析网络层级,并在心肌病模型中成功筛选出改善心脏收缩的治疗靶点。

引言
当前痛点:首先是数据饥渴,传统方法需海量转录组数据构建基因网络,罕见病及难取样组织研究受阻。其次是技术局限,现有模型无法跨任务复用知识,单任务训练效率低。
破局思路:迁移学习带来启发,借鉴NLP/CV领域,通过大规模预训练模型(如BERT)提取通用特征,微调适配下游任务。此外,单细胞技术揭示细胞异质性,为网络推断提供高精度数据基础。
文章目标:突破传统基因网络研究对海量数据的依赖,解决罕见病和难取样组织中因数据稀缺导致的机制解析与治疗靶点发现难题,实现小样本下的精准基因网络预测。
Geneformer方法概述
Geneformer是首个基于单细胞转录组的预训练迁移学习模型。采用Transformer架构,通过自监督学习从3000万单细胞数据中提取基因互作规律。通过“预训练-微调”范式,将大规模通用知识迁移至小样本任务,预测基因剂量敏感性、染色质动态和网络层级关系。
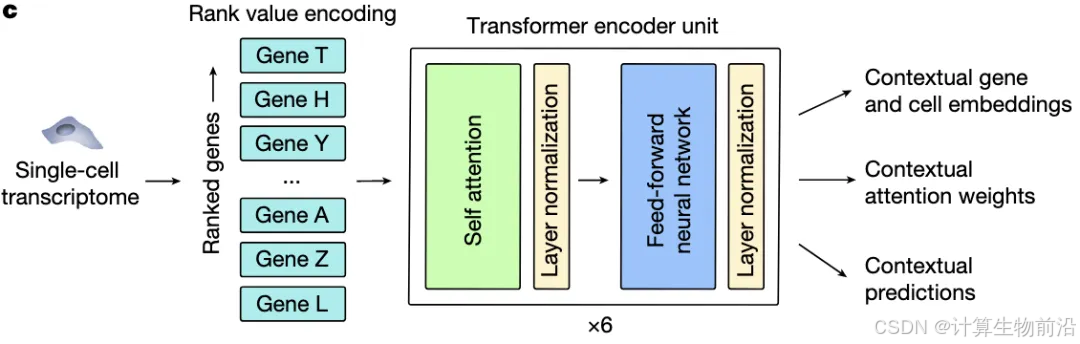
核心流程三步走
1. 数据预处理
-
构建Genecorpus-30M数据库,整合30种组织的单细胞数据。
-
独创基因排名编码:按细胞内表达量排序,消除技术偏差(如:高表达管家基因自动降权)。
2. 自监督预训练
-
任务设计:随机遮蔽15%基因,模型根据上下文预测缺失基因(类似“完形填空”)。
-
模型输出:生成基因和细胞的动态嵌入表示,编码网络层级信息。
3. 下游任务微调
-
添加轻量任务层(如分类层),用少量数据适配疾病建模、染色质状态预测等场景。
-
支持虚拟基因编辑:删除/激活目标基因,预测其对细胞状态的影响。
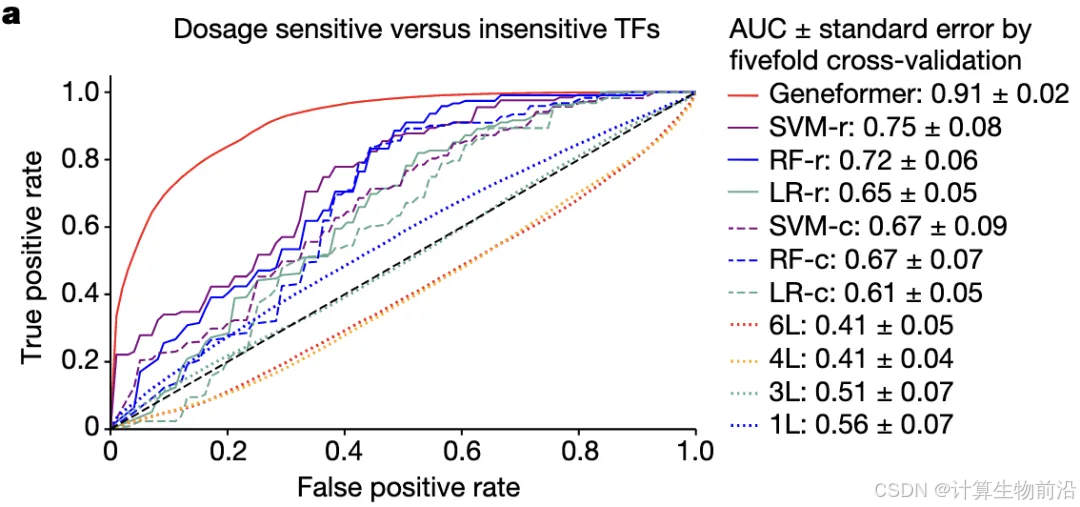
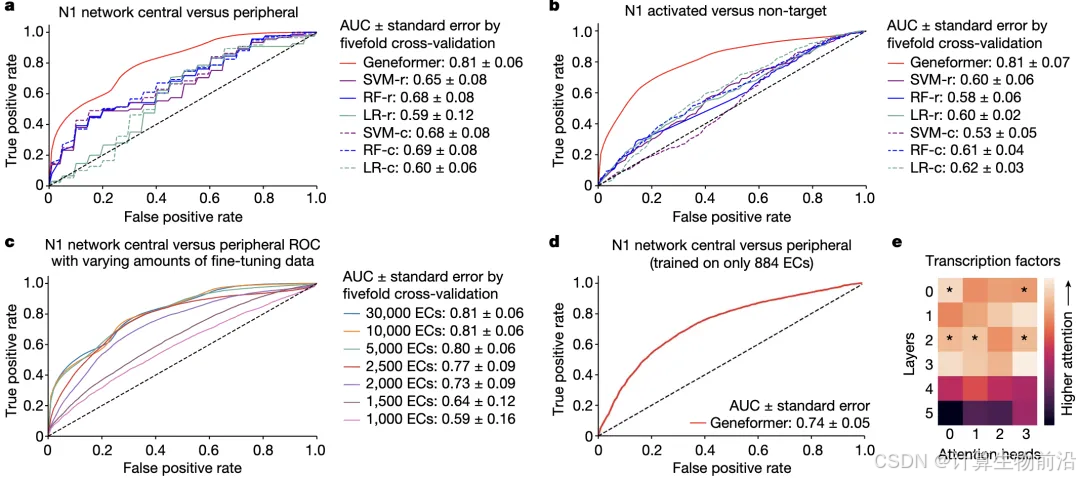
剂量敏感性预测
微调后模型区分剂量敏感/不敏感转录因子的AUC达0.91,成功预测22项研究中神经发育疾病相关基因的剂量敏感性(96%高置信基因与临床数据吻合)
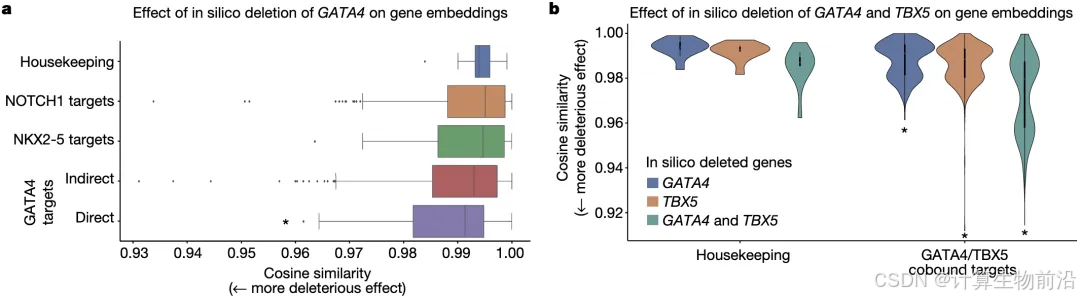
通过虚拟基因敲除发现,敲除心肌病相关基因(如GATA4、TBX5)显著改变心肌细胞基因表达网络(余弦相似度下降15%,p<0.05),且直接靶基因受影响程度是间接靶点的2.3倍
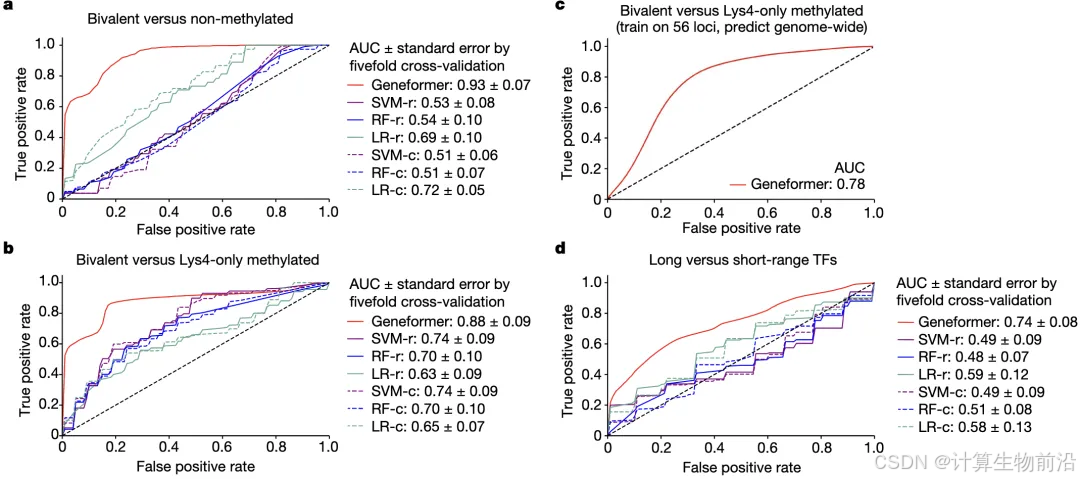
染色质状态预测突破
仅用56个保守区域的染色质标记数据微调,即可在全基因组范围预测双价染色质(H3K4me3/H3K27me3共标记)状态(AUC 0.78),准确识别MEF2等长程调控因子
网络层级自动解析
模型通过自注意力机制识别NOTCH1网络核心节点(中央基因注意力权重提升40%),并发现GATA4-TBX5协同调控靶点(共敲除效应超单基因之和1.8倍,p<0.01)
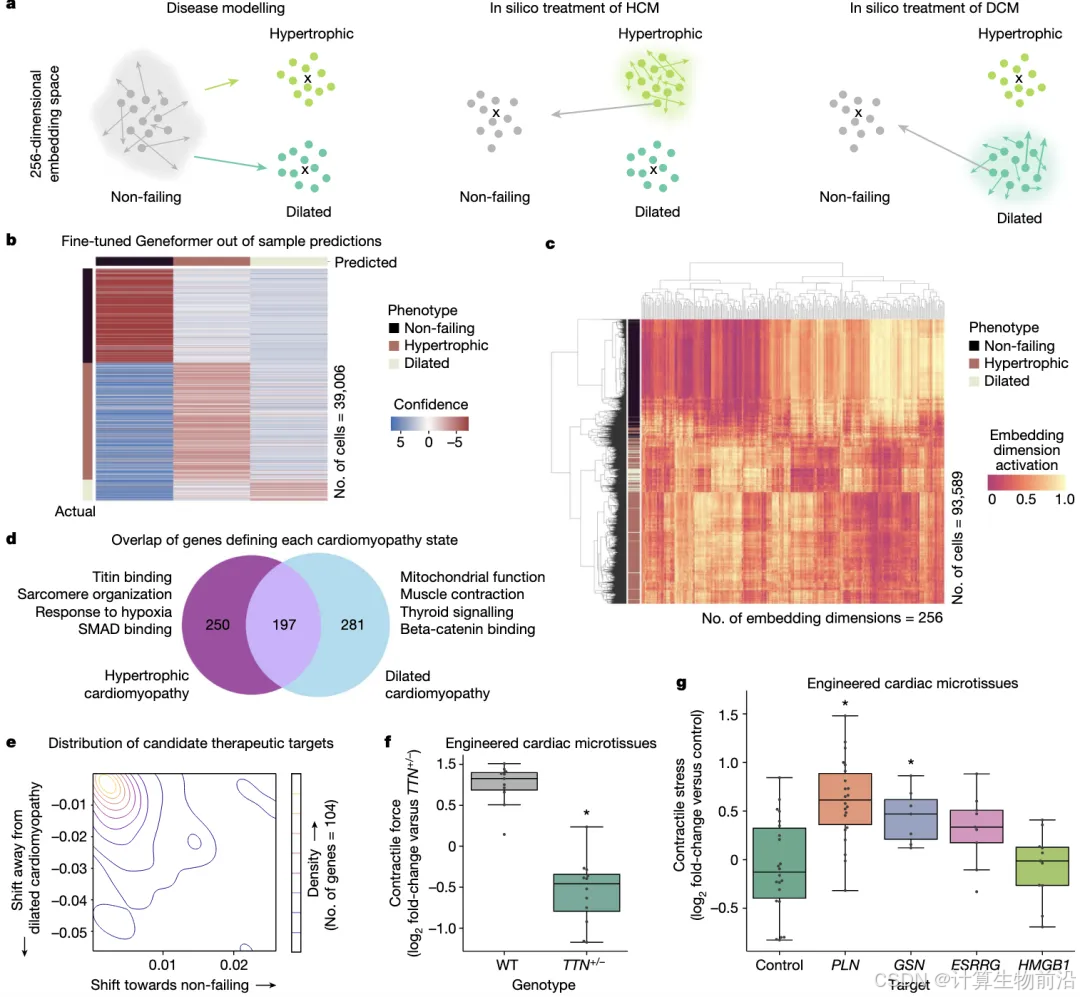
疾病靶点实验验证
在扩张型心肌病模型中,虚拟筛选发现抑制GSN(凝溶胶蛋白)和PLN(受磷蛋白)可逆转TTN突变心肌细胞收缩缺陷,CRISPR实验证实其敲除使收缩力提升42%(p<0.05)
参考资料
Theodoris, C.V., Xiao, L., Chopra, A. et al. Transfer learning enables predictions in network biology. Nature 618, 616–624 (2023). https://doi.org/10.1038/s41586-023-06139-9
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容






所有评论(0)